
在精准医学时代,NGS已经迅速成为检测多种体细胞变异、诊断疾病以及预测疗效的手段之一,然而分析验证过程仍然具有挑战性。
如果实验室希望分析患者材料,就必须优化和验证其工作流程,包括确定准确的检测极限。
正是由于对NGS肿瘤检测的日益普及,已经建立了多个检测肿瘤变异的具体指南,这些指南建议,样本应具有足够的序列数据,最小平均覆盖率为500X,以便能够可靠地检测出低至5%的等位基因突变频率。然而,随着新技术进步提升在低等位基因频率和覆盖偏差问题上的应用愈发重要,我们是否应该更进一步提高检测极限?
日益重要的理论检测极限
为了能够发现和诊断疾病,有必要验证NGS检测的最低等位基因频率。
尤其当考虑到不断发展的趋势并推动更高的测序准确性时,比如包括(但不限于):
• 检测极低水平下游离细胞DNA(cfDNA)的需要
• 含有大量肿瘤-正常DNA的异质组织样本
• 无创产前分析
• 补偿样品中的不均匀覆盖分布(例如,富含GC的区域)
此外,NGS实验的理论检测极限受分子特异性(去重复)、模板量和覆盖深度等参数的影响。
建库准备工作的选择对其LOD的影响
基于扩增子
在传统的扩增子组合中,使用相同序列的正向和反向引物生成短的扩增子,以特定区域为目标(如下图所示灰色“reads”)。
这些相同的末端序列使得在PCR扩增过程中很难从那些“复制的”分子中分离出独特的分子。理想情况下,只有独特的分子才能被用来计算等位基因的突变频率,以避免DNA扩增错误和偏差。
基于捕获
相反,基于捕获建库则(下图中)克服了这个问题,因为DNA首先被随机剪切,生成可以消除歧义或消除重复的随机起始/终止位点。但是,基于捕获建库的缺点是所需的模板量通常较高,可能不适合于来源困难的肿瘤。

不管建库方法如何,使用已知的参考标准品测定实验的检测限通常被认为是良好的做法。
考虑到覆盖偏差
重要的是,由于样本的覆盖分布不均匀,可能需要提高测序深度,以实现目标区域的覆盖。
测序覆盖深度也在检测极限中起作用。如果你不受样本材料的限制,那么下面的方程式的分母不受分子的限制,而是变成Reads数决定的:
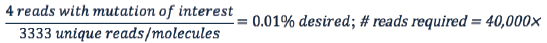
同样,假设需要四个包含目标突变序列的读数来检测0.01%或更低的等位基因频率,那么这个区域的测序深度必须至少为40000X。
重要的是,由于样本的覆盖分布不均匀,可能需要进行更深入的测序,以实现目标区域40000×的覆盖。当把实验噪声考虑在内时,实际的检测极限可能会高得多。
覆盖偏差示例
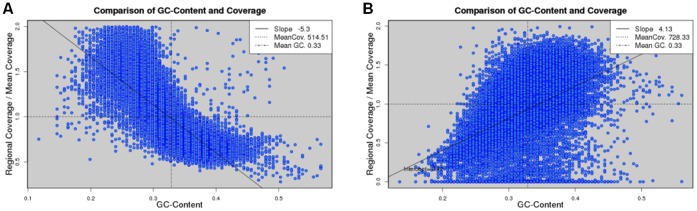
图- Chen et al
数据集来源于金黄色葡萄球菌USA300(A)和金黄色葡萄球菌mrsa252 基因组(B)。读值覆盖深度是标准化的两个平均值,代表水平线。垂直线A 表示平均GC含量。基于数据点的直线和斜率定义为“GC偏差程度。两个用例分别代表负面和正面的GC的偏差。
结论
推动NGS理论检测极限变得越来越重要,在实施到临床工作流程之前,必须用已知的标准评估方法进行性能验证。
为了能够发现和诊断疾病,有必要验证NGS实验可能的最低等位基因频率检测能力。
相比组织检测,液体活检具有方便、快速、即时、便宜、微创的特点,这些特点使其迅速成为目前肿瘤基因检测甚至是肿瘤早筛的研究热门。
菁良基因肺癌ctDNA标准品套装除了日常质控您的检测流程外,其低至0.1%的突变频率还可以帮助临床实验室根据本身实际情况,进行最低检测限的验证。

![]()
菁良ctDNA标准品套装
适用的液体活检各过程:ctDNA提取,变异检测,信息分析;
适用的检测方法:NGS测序,PCR检测等。
参考文献
New York State Department of Health. (2014, January). "Next Generation" Sequencing (NGS) guidelines for somatic genetic variant detection.



